Ưu điểm, đồng thời là nhược điểm của CRISPR
CRISPR được phát hiện lần đầu tiên ở trong vi khuẩn và vi khuẩn cổ năm 1987 [1]. Sau khi được phát hiện, giải trình tự gene của CRISPR cho thấy nó tương đồng với trình tự của vi khuẩn và plasmid; đồng thời nằm cạnh gene cas - gene mã hóa cho enzyme nuclease xúc tác việc cắt DNA ngoại sinh. Do đó, họ cho rằng các chuỗi CRISPR có thể là một phần của một hệ thống phòng thủ chống lại sự xâm nhập của các tác nhân virus và các plasmid, gọi là phức hợp CRISPR-Cas. Năm 2012, lần đầu tiên công nghệ CRISPR được áp dụng làm kỹ thuật chỉnh sửa hệ gene nhờ công trình nghiên cứu của nhóm tác giả Jennifer Doudna và Emmanuelle Charpentier tại Trường Đại học California, Hoa Kỳ [2]. Trong nghiên cứu này, nhóm tác giả đã đưa vào trong tế bào một phức hệ bao gồm enzyme Cas9 nuclease và RNA hướng dẫn (guide RNA) tự thiết kế để cắt đoạn DNA tại những vị trí mong muốn. Năm 2015, Tạp chí Science bầu chọn CRISPR là công nghệ khoa học quan trọng nhất, khởi đầu kỷ nguyên công nghệ sinh học mới, giúp chỉnh sửa thông tin di truyền của mọi tế bào một cách nhanh chóng và chính xác [3]. Chính vì thế, các nhà khoa học đã ví CRISPR/Cas9 là “chiếc kéo phân tử”, có khả năng cắt dán chính xác các đoạn gene, nhờ đó chỉnh sửa, loại bỏ các gene đột biến, gene gây bệnh.
Hình 1. Nguyên lý hoạt động của CRISPR/Cas9 (Nguồn: Nature News).
Hệ thống CRISPR/Cas9 bao gồm 2 thành phần: enzyme Cas9 nuclease và RNA hướng dẫn. Nhờ đoạn trình tự bổ sung của RNA hướng dẫn tới trình tự đích mà phức hợp này có thể tìm thấy vị trí cần chỉnh sửa trên hệ gene. Khi phức hợp Cas9 và RNA hướng dẫn bám vào trình tự đích, enzyme Cas9 sẽ cắt đoạn DNA trên cả 2 chuỗi. Khi cả 2 chuỗi DNA bị cắt đứt, tế bào sẽ sửa chữa bằng cách chèn hoặc xóa thêm các nucleotide. Ở khía cạnh tích cực, đặc tính này sẽ được tận dụng để loại bỏ các gene gây bệnh. Tuy nhiên, chính sự tạo ra đứt gãy sợi đôi lại là một hành động phá hoại bộ gene. Khi tế bào cố gắng sửa chữa, nó có thể xóa hoặc chèn ngẫu nhiên nucleotide một cách thiếu chính xác (gọi là đột biến điểm Indels). Điều này có thể gây ra những đột biến không mong muốn, thậm chí nguy hiểm. Đây chính là điểm yếu giới hạn ứng dụng của CRISPR/Cas9 trong nghiên cứu cơ bản và nông nghiệp [4, 5].
“Prime editing” - sự cải tiến mới của CRISPR
Trong CRISPR tiêu chuẩn, enzyme Cas9 được sử dụng để cắt trên cả 2 mạch của phân tử DNA. Do đó, CRISPR cực kỳ hữu ích để phá vỡ gene (chèn hoặc xóa) trên đoạn DNA lớn. Tuy nhiên, rất khó để sử dụng hệ thống tiêu chuẩn này để thực hiện các chỉnh sửa tinh vi hơn, chẳng hạn như thay đổi base T thành base A. Do đó, kỹ thuật chỉnh sửa base (Base editing/BE) được ra đời để giải quyết bài toán này. Kỹ thuật này chỉnh sửa base trực tiếp bằng cách chuyển một cặp base của DNA thành một cặp khác mà không làm đứt phân tử acid nucleic. Ưu điểm của phương pháp này là ít gây đột biến Indels và có hiệu quả chỉnh sửa cao hơn [5]. Kỹ thuật này lần đầu được phát triển bởi nhà sinh học David Liu và cộng sự tại Đại học Harvard [6]. Hạn chế của phương pháp này là bị tác động giới hạn các cặp base nhất định (mới chỉ hiệu quả khi hoán đổi C-G thành T-A), ít có hiệu quả trên các đột biến đa điểm (như bệnh đột tử do tim/SCD, bệnh lý huyết học ác tính…) và thường gây ra đột biến lệch mục tiêu.
Mới đây nhất, các nhà khoa học thuộc Viện Broad của MIT và Harvard (The Broad Institute of MIT and Harvard - một trung tâm nghiên cứu bộ gene và y sinh đặt tại Cambridge, Massachusetts, Hoa Kỳ) đã nghiên cứu nghiên cứu phát triển một công cụ chỉnh sửa gene mới có tên là Prime editing (PE). Nhóm nghiên cứu đã thử nghiệm các biến thể protein mà họ đã chỉnh sửa với PE trên một số tế bào của người và chuột [7]. PE thể hiện lợi thế rõ ràng so với 2 công cụ chỉnh sửa bộ gene nêu trên ở khả năng chèn và xóa chính xác đến từng base vào bộ gene mà không cần tạo ra DSB (đứt gãy cả 2 chuỗi polynucleotit/đứt gãy kép/double strand break) [8]. Theo đó, PE khắc phục thiếu sót của cả hai phương pháp trên bằng cách cải tiến cả RNA hướng dẫn và enzyme cắt Cas9. PE có thể hoán đổi chính xác và hiệu quả ở bất kỳ cặp base đơn lẻ nào. Hệ thống này bao gồm một phiên bản sửa đổi của enzyme Cas9 được hợp nhất với một enzym khác (enzym phiên mã ngược) và một RNA hướng dẫn được thiết kế đặc biệt gọi là pegRNA. Phần sau của PE chứa bản chỉnh sửa gene mong muốn và giúp điều khiển toàn bộ hệ thống chỉnh sửa đến một vị trí cụ thể trên DNA của tế bào. Khi đến đích, Cas9 chỉ cắt một sợi đơn của chuỗi xoắn kép DNA, thay vì cả hai. Enzym phiên mã ngược sử dụng đoạn mồi để bắt đầu phiên mã thông tin di truyền được mã hóa trong pegRNA, sau đó thay thế vào vị trí được đánh dấu (giống như chức năng tìm kiếm và chỉnh sửa trên phần mềm MS Word). Quá trình này sẽ được kết thúc khi hệ thống chỉnh sửa chính yêu cầu tế bào làm lại chuỗi DNA khác để khớp với thông tin di truyền mới. Trên lý thuyết, PE có thể sửa chữa khoảng 89% trong số hơn 75.000 biến thể di truyền đã được tìm thấy có liên quan đến các bệnh tật ở người [7].
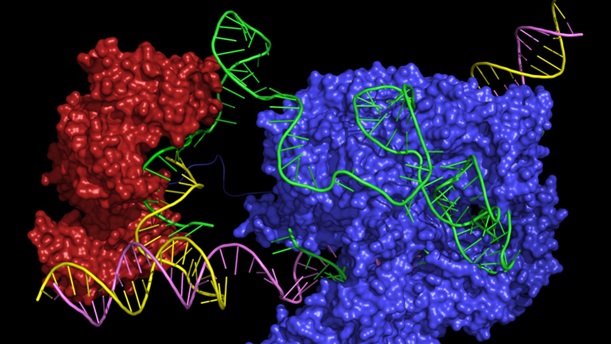
.jpg)
Hình 2. Các thành phần của Prime editing (PE). PE kết hợp 2 enzyme Cas9 (xanh dương) và enzyme phiên mã ngược (màu đỏ) với một RNA hướng dẫn (màu đỏ) để đưa cả phức hệ đến một vị trí DNA mạch đôi cụ thể (vàng và tím), đồng thời mang luôn trình tự muốn chèn vào vị trí này (Nguồn: science.org).
Thách thức ở phía trước
Một trong những mục tiêu chính của bất kỳ kỹ thuật chỉnh sửa gene nào là tính hiệu quả, bởi nó quy định chiến lược tiếp cận lâm sàng. Một nghiên cứu do Sürün dẫn đầu đã chứng minh rằng, PE hoạt động hiệu quả (3-6%) trong tế bào iPS (tế bào gốc đa năng) của người [9]. Trong một nghiên cứu khác, Rousseau và cộng sự cũng báo cáo việc sử dụng BE để tạo ra đột biến Ala673Thr gây bệnh Alzheimer trong gene APP thành công [10]. Ngược lại, PE cho phép họ tạo chính xác đột biến Ala673Thr, mặc dù hiệu quả chỉnh sửa thấp hơn nhiều so với BE. Đáng chú ý, họ báo cáo thêm rằng lần PE thứ hai có thể được sử dụng để tăng hiệu quả chỉnh sửa [10]. Yếu tố đóng vai trò là nút thắt cổ chai trong việc ứng dụng các công nghệ gene trên lâm sàng nằm ở việc dự đoán và đánh giá các tác động ngoài mục tiêu. Việc không hình thành DSB (đứt gãy cả hai chuỗi polynucleotit/đứt gãy kép/double strand break) hứa hẹn mang lại tính an toàn đáng kể cho các liệu pháp dựa trên PE. Trong 2 thí nghiệm độc lập, Schene và cộng sự đã không phát hiện được bất kỳ chỉnh sửa nào ngoài mục tiêu sau khi thực hiện phân tích trình tự toàn bộ bộ gene [11]. Ngoài ra, PE không chỉ chứng minh các hiệu chỉnh hiệu quả cao ở nhiều loại tế bào mà còn cho thấy tỷ lệ đột biến indel rất thấp. Điều này đặc biệt quan trọng đối với in vivo. Liu và cộng sự đã sử dụng hệ thống PE để sửa chữa có hiệu quả một đột biến trong gene Hoxd13 trong phôi chuột, một tế bào tương đồng với một gene có quan hệ lâm sàng ở người [12]. Thật đáng khích lệ, các dữ liệu cho thấy PE hoạt động trong nhiều loại tế bào, tế bào cơ quan và phôi chuột. Điều thú vị là cũng có những dấu hiệu cho thấy việc chỉnh sửa phôi có thể sử dụng những khác biệt nhỏ trong các con đường sửa chữa DNA [13]. Dĩ nhiên, PE vẫn có những hạn chế nhất định. Đầu tiên, 11% các đột biến gây bệnh đã biết là do thay đổi số lượng bản sao của gene. Không rõ liệu PE có thể chèn hoặc loại bỏ các DNA này hay không, khi mà chiều dài đầy đủ của chúng có thể lên tới 2,4 triệu base. Thứ hai, BE và CRISPR tiêu chuẩn hiện nay đã được thử nghiệm kỹ lưỡng hơn nhiều so với PE trên nhiều loại tế bào và mô hình động vật khác nhau. Những công nghệ chỉnh sửa trước đó cũng có thể hiệu quả hơn cho một số mục đích nhất định, vì vậy chúng vẫn có thể sẽ hữu ích trong y sinh học. Đối với PE, vẫn cần có thêm nhiều nghiên cứu bổ sung trước khi có thể xem xét triển khai các thử nghiệm lâm sàng trên người. Đặc biệt là tính an toàn và hiệu quả của công nghệ này đối với nhiều loại tế bào và tiềm năng của nó trong việc chỉnh sửa chính xác và an toàn các gene ở mô được nhắm mục tiêu bên trong động vật sống và con người.
TÀI LIỆU THAM KHẢO
[1] Y. Ishino, H. Shinagawa, K. Makino, M. Amemura, A. Nakata (1987), “Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product”, ASM Journals, Journal of Bacteriology, 169, pp.12, DOI: 10.1128/jb.169.12.5429-5433.1987.
[2] Martin Jinek, Krzysztof Chylinski, Ines Fonfara, Michael Hauer, Jennifer A. Doudna and Emmanuelle Charpentier (2012), “A programmable dual-RNA-Guided DNA endonuclease in adaptive bacterial immunity”, Science, 337(6096), pp.816-821, DOI: 10.1126/science.1225829.
[3] https://www.science.org/content/article/and-science-s-2015-breakthrough-year.
[4] https://www.science.org/content/article/new-prime-genome-editor-could-surpass-crispr.
[5] N. Gaudelli, A. Komor, H. Rees, et al. (2017), “Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage”, Nature, 551, pp.464-471, DOI: 10.1038/nature24644.
[6]https://news.harvard.edu/gazette/story/2021/12/new-technique-enables-manipulation-of-large-dna-segments/
[7] https://directorsblog.nih.gov/tag/pegrna/.
[8] G. Zhang, Y. Liu, S. Huang, et al. (2022), “Enhancement of prime editing via xrRNA motif-joined pegRNA”, Nat Commun, 13, p.1856, DOI: 10.1038/s41467-022-29507-x.
[9] D. Sürün, A. Schneider, J. Mircetic, K. Neumann, F. Lansing, M. Paszkowski-Rogacz, et al. (2020), “Efficient generation and correction of mutations in human iPS cells utilizing mRNAs of CRISPR base editors and prime editors”, Genes, 11, p.511, DOI: 10.3390/genes11050511.
[10] https://www.biorxiv.org/content/10.1101/2020.07.31.230565v1.
[11] I.F. Schene, et al. (2021), “Prime editing for functional repair in patient-derived disease models”, Nat. Commun, 11, p.5352.
[12] Y. Liu, et al. (2020), “Efficient generation of mouse models with the prime editing system”, Cell Discov, 6, pp.1-4.
[13] J. Scholefield, P.T. Harrison (2021), “Prime editing - an update on the field”, Gene Ther, 28, pp.396-401, DOI: 10.1038/s41434-021-00263-9